把 169861 個(gè)生物物種數(shù)據(jù)裝進(jìn)大模型,大模型竟 get 到了生物中心法則的奧秘——
不僅能識(shí)別 DNA、RNA 與相應(yīng)蛋白質(zhì)之間的內(nèi)在聯(lián)系,在基因分類、蛋白質(zhì)相互作用預(yù)測(cè)、熱穩(wěn)定性預(yù)測(cè)等 7 種不同類型任務(wù)中也能比肩 SOTA 模型。
模型名為LucaOne,由阿里云飛天實(shí)驗(yàn)室生物智能計(jì)算團(tuán)隊(duì)打造。
相比 AlphaFold 3 因未開源遭到 650 多名學(xué)者聯(lián)名批評(píng),LucaOne 訓(xùn)練推理代碼及相關(guān)數(shù)據(jù)目前均已開源。
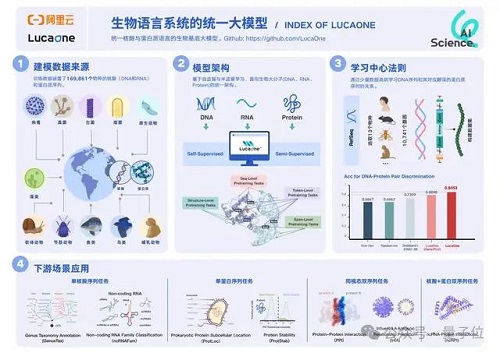
LucaOne 是目前首個(gè)全生物系統(tǒng)的核酸語(yǔ)言 + 蛋白語(yǔ)言的融合基座模型。換句話說,LucaOne 由核酸(DNA、RNA)和蛋白質(zhì)序列聯(lián)合訓(xùn)練而來。
通過一系列實(shí)驗(yàn),研究人員發(fā)現(xiàn)它能廣泛適用各種下游任務(wù)。
在含 13 個(gè)物種、關(guān)系對(duì)總數(shù)量為 24000 的核酸序列和其對(duì)應(yīng)蛋白的正負(fù)樣本數(shù)據(jù)集中,LucaOne 提供表征的模型達(dá)到 0.85 的預(yù)測(cè)準(zhǔn)確率。
遠(yuǎn)高于目前業(yè)內(nèi)最好的預(yù)訓(xùn)練模型組合 ESM-3B+DNAbert2(0.73)及其他建模方式,也顯著高于 LucaOne 的單核酸訓(xùn)練版本 + 單蛋白訓(xùn)練版本。
其他任務(wù)如針對(duì)流感 H3N2 病毒疫苗有效性(免疫逃逸風(fēng)險(xiǎn))的預(yù)測(cè),LucaOne準(zhǔn)確率可達(dá) 100%。
量子位也聯(lián)系到了論文一作,聊了聊 LucaOne 的實(shí)現(xiàn)細(xì)節(jié),以及 AI for Science 在生物科學(xué)領(lǐng)域的發(fā)展。

核酸和蛋白質(zhì)序列聯(lián)合訓(xùn)練
總的來說,LucaOne 圍繞中心法則的數(shù)據(jù)進(jìn)行構(gòu)建,能夠?qū)W習(xí)到中心法則背后的原理和邏輯,可提取基因轉(zhuǎn)錄和蛋白質(zhì)翻譯過程中固有的復(fù)雜模式和關(guān)系,在使用層面相當(dāng)于提供了一個(gè)對(duì) DNA、 RNA、蛋白質(zhì)的無(wú)差別表征。
PS:
分子生物學(xué)的中心法則即遺傳信息從 DNA 傳遞給 RNA,再?gòu)?RNA 傳遞給蛋白質(zhì)的過程,這一過程包括 DNA 的復(fù)制、RNA 的轉(zhuǎn)錄和蛋白質(zhì)的翻譯。
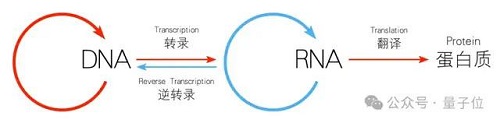
展開來看,LucaOne 整個(gè)工作流是這樣?jì)饍旱模?/p>
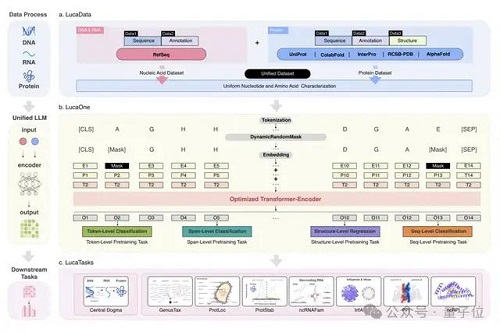
從技術(shù)上講,構(gòu)建 LucaOne 的難點(diǎn)首先是數(shù)據(jù)集的構(gòu)建。
在生命科學(xué)領(lǐng)域,真實(shí)存在的只是分子數(shù)據(jù)。
例如,核酸的表示方式是 4 種堿基。DNA 是腺嘌呤(A)、鳥嘌呤(G)、胞嘧啶(C)和胸腺嘧啶(T);RNA 是腺嘌呤(A)、鳥嘌呤(G)、胞嘧啶(C)、尿嘧啶(U)。蛋白質(zhì)由氨基酸組成,自然界存在的氨基酸大約有 20-22 種,每種氨基酸也用一個(gè)字母表示。
而人類為理解這些分子的性質(zhì)與作用,通常需要添加很多注釋信息,包括一些圖片的注釋。注釋信息屬于人類語(yǔ)言,自然界本身不存在,從而就形成了一種生命科學(xué)領(lǐng)域的從 " 自然界 " 語(yǔ)言到 " 人類文化 " 語(yǔ)言的跨模態(tài)。
因此,LucaOne 的預(yù)訓(xùn)練數(shù)據(jù)不僅包含 DNA、RNA、蛋白質(zhì)這三類分子的序列(核苷酸序列或者氨基酸序列)數(shù)據(jù),同時(shí)還使用了這些分子的注釋信息。
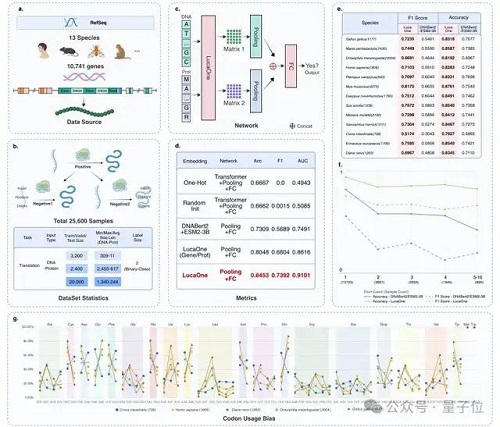
總共涵蓋了 169861 個(gè)物種的核酸和蛋白質(zhì)序列和注釋信息,分為兩部分:
核酸數(shù)據(jù)集來自 RefSeq,包括核酸序列及注釋;蛋白質(zhì)數(shù)據(jù)集來自 InterPro、UniProt、ColabFold、RCSB-PDB、AlphaFold2,包括蛋白質(zhì)序列、注釋和三維結(jié)構(gòu)。
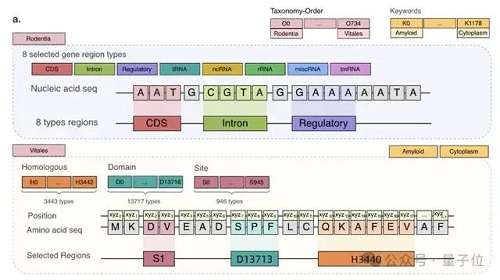
據(jù)介紹,在數(shù)據(jù)集的收集處理方面,阿里云飛天實(shí)驗(yàn)室與中山大學(xué)、浙江大學(xué)等多個(gè)團(tuán)隊(duì)進(jìn)行了合作。
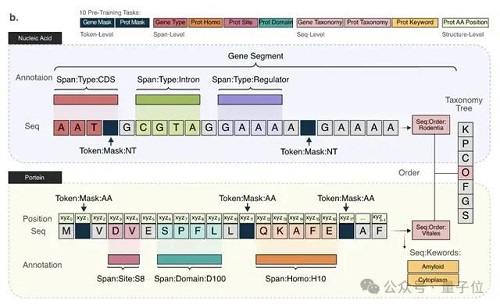
另一大難點(diǎn)是生物分子序列的預(yù)測(cè)和大語(yǔ)言模型預(yù)測(cè)下一個(gè) token 不同,在模型訓(xùn)練階段還需要一些專門的設(shè)計(jì)。
LucaOne 采用了Transformer-Encoder架構(gòu),由 20 個(gè)編碼器塊組成,嵌入維度為 2560,總參數(shù)量1.8B。
研究人員在此基礎(chǔ)上進(jìn)行了一些優(yōu)化:
使用 Pre-Layer 歸一化代替 Post-Layer 歸一化,以便更好地訓(xùn)練深層網(wǎng)絡(luò);使用旋轉(zhuǎn)位置嵌入(RoPE)代替?zhèn)鹘y(tǒng)絕對(duì)位置編碼,以推理更長(zhǎng)序列。
此外,在數(shù)據(jù)處理和模型訓(xùn)練過程中,核苷酸和氨基酸用統(tǒng)一的方式進(jìn)行表征或編碼。通過 token-type embeddings 實(shí)現(xiàn)核酸和蛋白質(zhì)序列的混合訓(xùn)練,區(qū)分核苷酸(0)和氨基酸(1)。
在兩個(gè)自監(jiān)督掩碼任務(wù)的基礎(chǔ)上,研究人員還增加了八個(gè)半監(jiān)督預(yù)訓(xùn)練任務(wù),通過序列注釋增強(qiáng)模型對(duì)數(shù)據(jù)的理解。
已能理解基因和蛋白對(duì)應(yīng)關(guān)系
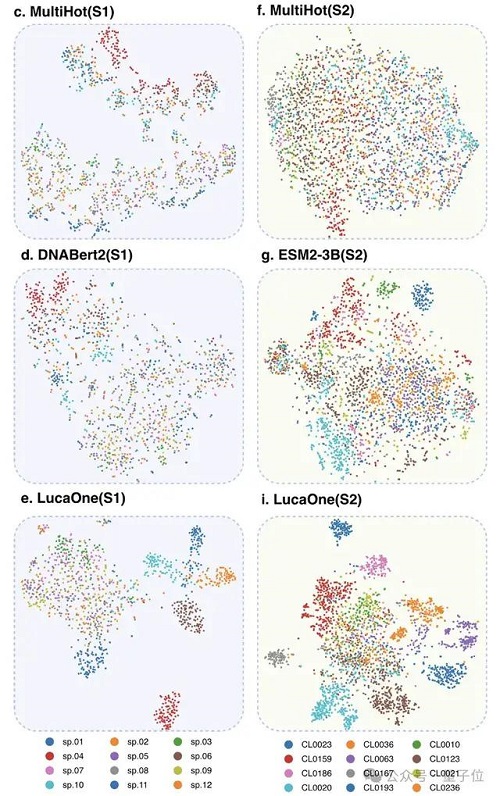
為驗(yàn)證核酸和蛋白質(zhì)數(shù)據(jù)混合訓(xùn)練的優(yōu)勢(shì),研究人員分別使用核酸和蛋白質(zhì)數(shù)據(jù)單獨(dú)訓(xùn)練了兩個(gè)額外的模型—— LucaOne-Gene 和 LucaOne-Prot,并使用相同的 5.6M checkpoint 在分子生物學(xué)中心法則任務(wù)中進(jìn)行了比較。
使用 t-SNE 可視化說明,與其他模型相比,LucaOne 的嵌入在兩個(gè)數(shù)據(jù)集上呈現(xiàn)出更緊密的聚類,可能包含了更多上下文信息。
為驗(yàn)證 LucaOne 通過廣泛學(xué)習(xí)基因及蛋白語(yǔ)言,已具備對(duì)生物學(xué)中心法則里的基因和蛋白對(duì)應(yīng)關(guān)系的理解能力,研究人員設(shè)計(jì)了一個(gè)數(shù)據(jù)集及評(píng)測(cè)任務(wù)。
選取 13 個(gè)物種的核酸序列和其對(duì)應(yīng)蛋白的正負(fù)樣本數(shù)據(jù)集,關(guān)系對(duì)總數(shù)量為 24000,其中正負(fù)樣本比例 1:2。基因序列數(shù)據(jù)是其在基因組的原始數(shù)據(jù),包括了大量的非編碼區(qū)(內(nèi)含子,調(diào)控元件,及 " 垃圾片段 " 等)。
采用訓(xùn)練:驗(yàn)證:測(cè)試比例為:4:3:25;即僅 3200 組數(shù)據(jù)作為訓(xùn)練,18750 組數(shù)據(jù)作為測(cè)試集來預(yù)測(cè)其核酸序列是否可以翻譯成數(shù)據(jù)組里的蛋白序列。
結(jié)果 LucaOne 提供表征的模型達(dá)到0.85的預(yù)測(cè)準(zhǔn)確率,不僅遠(yuǎn)高于目前業(yè)內(nèi)最好的預(yù)訓(xùn)練模型組合 ESM-3B+DNAbert2(0.73)及其他建模方式,也明顯高于 LucaOne 的單核酸訓(xùn)練版本 + 單蛋白訓(xùn)練版本。
這表明這兩種大分子數(shù)據(jù)聯(lián)合訓(xùn)練可以顯著增強(qiáng)模型的學(xué)習(xí)效果。
有意思的是,研究人員發(fā)現(xiàn)在模型細(xì)分表現(xiàn)里,LucaOne海鞘這種生物里的預(yù)測(cè)表現(xiàn)比較差(其他模型也類似),進(jìn)一步分析海鞘的特性表明,因?yàn)檫M(jìn)化適應(yīng)性等各種原因,海鞘利用中心法則的具體規(guī)則 - 密碼子偏好性,和其他生物明顯不同。
他們猜測(cè) LucaOne 可能用的是另一種中心法則語(yǔ)法" 方言 ",而這種 " 方言 " 在訓(xùn)練數(shù)據(jù)集里僅有 100 條,因此模型沒有很好的學(xué)習(xí)到這種規(guī)則。
在其他下游任務(wù)中,LucaOne 對(duì)不同類型輸入的下游任務(wù)也廣泛適用。
具體來說,研究人員評(píng)估了 7 個(gè)不同類型的下游生物計(jì)算任務(wù),包括 :
單序列任務(wù):GenusTax(屬分類)、ncRNAFam(ncRNA 家族分類)、ProtLoc(蛋白質(zhì)亞細(xì)胞定位)、ProtStab(蛋白質(zhì)熱穩(wěn)定性預(yù)測(cè))。同源序列對(duì)任務(wù):InfA(流感血凝素分析)、PPI(蛋白質(zhì)相互作用預(yù)測(cè))。異源序列對(duì)任務(wù):ncRPI(ncRNA- 蛋白質(zhì)相互作用預(yù)測(cè))。
為簡(jiǎn)化下游任務(wù),研究人員使用了三種對(duì)應(yīng)不同輸入形式的簡(jiǎn)單網(wǎng)絡(luò)架構(gòu):
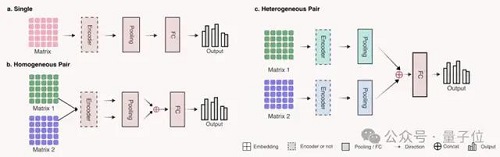
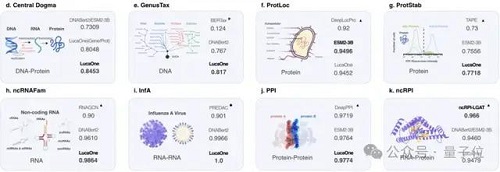
結(jié)果表明,GenusTax、ProtStab、ncRNAFam、InfA、PPI 任務(wù)上,LucaOne 顯著優(yōu)于其他模型;ProtLoc 任務(wù)上,LucaOne 與 ESM2-3B 相當(dāng),優(yōu)于 SOTA;ncRPI 任務(wù)上,LucaOne 優(yōu)于 DNABert2+ESM2-3B 的組合:
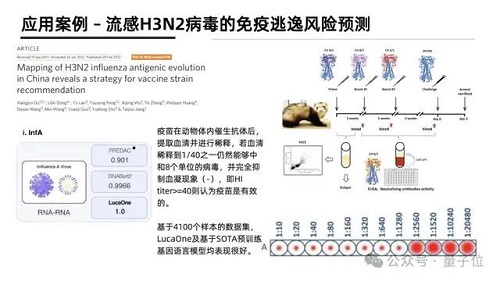
值得一提的是,在流感 H3N2 病毒的免疫逃逸風(fēng)險(xiǎn)預(yù)測(cè)中,研究人員采用了 1968 年至 2010 年間分離的大規(guī)模 H3N2 病毒 HA 序列數(shù)據(jù)進(jìn)行了基于流感毒株抗原關(guān)系的預(yù)測(cè)模型。
通過病毒 HA 抗原序列來預(yù)測(cè)其是否會(huì)誘導(dǎo) HIA 實(shí)驗(yàn)的血凝現(xiàn)象,進(jìn)而預(yù)測(cè)其是否在特定人群中會(huì)發(fā)生免疫逃逸。
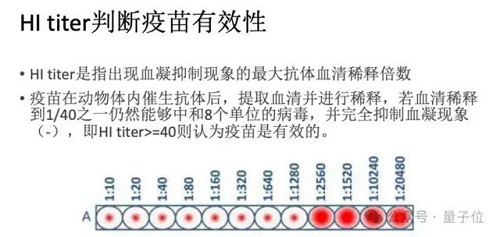
要知道,目前預(yù)防與控制流感最有效的方法是接種流感疫苗,但是由于流感病毒極快的變異速度,導(dǎo)致不能及時(shí)與準(zhǔn)確地推薦與流行病毒相匹配的流感疫苗株。根據(jù) WHO 和 CDC 的監(jiān)測(cè),流感疫苗的有效性在 40%-60% 之間。因此準(zhǔn)確預(yù)測(cè)流行毒株,判斷免疫逃逸風(fēng)險(xiǎn)是一個(gè)重要且困難的命題。
研究人員使用基于 LucaOne+ 一層感知機(jī)的模型達(dá)到了 100% 的準(zhǔn)確率。
這也說明 LucaOne 學(xué)習(xí)的大量核酸序列信息,包括大量其他病毒序列,為計(jì)算特定任務(wù)提供了很好的信息補(bǔ)充。
更多細(xì)節(jié),感興趣的家人們可查看原論文。
"Science for AI"
如前文所述,LucaOne 背后開發(fā)團(tuán)隊(duì)來自阿里云飛天實(shí)驗(yàn)室 LucaTeam,LucaTeam 也與多個(gè)團(tuán)隊(duì)展開了深度合作。
中山大學(xué)醫(yī)學(xué)院施莽教授及其團(tuán)隊(duì)參與了 LucaOne 模型的數(shù)據(jù)設(shè)計(jì)與驗(yàn)證。施莽教授認(rèn)為:
LucaOne 是一項(xiàng)極為重要的嘗試。最讓我驚訝的是,在沒有任何先驗(yàn)知識(shí)的前提下,LucaOne 確實(shí)能夠更有效地學(xué)習(xí)中心法則中核酸與蛋白質(zhì)之間的對(duì)應(yīng)關(guān)系。
中國(guó)醫(yī)學(xué)科學(xué)院北京協(xié)和醫(yī)學(xué)院病原生物學(xué)研究所所長(zhǎng)、美國(guó)微生物科學(xué)院會(huì)士舒躍龍教授及其團(tuán)隊(duì)參與了 LucaOne 在流感病毒方面的分析與驗(yàn)證工作。舒躍龍教授表示:
將前沿的 AI 技術(shù)與病原生物學(xué)相結(jié)合具有重大的科學(xué)意義和社會(huì)價(jià)值。通過這種緊密的跨學(xué)科協(xié)作,我們能探索更多病原生物起源進(jìn)化、跨種傳播以及感染致病等方面的規(guī)律,為傳染病防控和生物安全做出更大的貢獻(xiàn)。
此外,論文一作賀勇是阿里云飛天實(shí)驗(yàn)室生物計(jì)算高級(jí)算法專家,我們也就 LucaOne 與其展開聊了聊 AI for Science 在生物科學(xué)領(lǐng)域的發(fā)展。
在他看來,AI for Science 在生物科學(xué)領(lǐng)域正處于剛起步的階段,考慮到 AI 可解釋性的問題,基本上現(xiàn)在他們還只是把 AI 當(dāng)做一個(gè)工具,但同時(shí)現(xiàn)在是發(fā)展 AI for Science 的一個(gè)很好的時(shí)間節(jié)點(diǎn)。
因?yàn)楝F(xiàn)在測(cè)序技術(shù)發(fā)展非常迅速,測(cè)序成本下降很多,分子序列數(shù)據(jù)很容易就能獲得。有了數(shù)據(jù)大量的積累,就可以數(shù)據(jù)驅(qū)動(dòng)的 AI for Science 相關(guān)工作。
不過,目前的局限在于測(cè)序得到還是分子的序列數(shù)據(jù),而真實(shí)世界每個(gè)分子的存在是一個(gè)空間結(jié)構(gòu),這可能就需要更復(fù)雜的模型來處理。
而不同學(xué)科間的研究方法不同、探索微觀世界也受限于目前的設(shè)備技術(shù),賀勇認(rèn)為人類對(duì)生物科學(xué)這個(gè)領(lǐng)域的認(rèn)識(shí)目前也只是冰山一角,還無(wú)法從全局角度構(gòu)建一個(gè)全面通用的系統(tǒng)。
最后他還補(bǔ)充道:
目前大家對(duì) AI for Science 越來越重視,相當(dāng)于用 AI 去解決具體的問題。我想接下來還應(yīng)該回過頭來看 Science 能為 AI 帶來什么?解決具體問題是第一步,最后通過問題反哺技術(shù)本身其實(shí)可能也是應(yīng)該考慮的。


